Classification Evaluation
Contents
Classification Evaluation#
분류 문제(binary/multi-class/multi-label)에서 데이터를 통해 생성한 모델이 좋은지 판단하기 위해 자주사용하는 평가 함수에 대해서 알아봅니다.
Accuracy score#
Accuracy score 는 실제 값과 예측 값이 일치하는 비율 또는 갯수를 나타냅니다.
\( \texttt{accuracy}(y, \hat{y}) = \frac{1}{n_\text{samples}} \sum_{i=0}^{n_\text{samples}-1} 1(\hat{y}_i = y_i) \)
이는 multi-label 분류 문제에서도 동일하게 사용됩니다.
import numpy as np
from sklearn.metrics import accuracy_score
# Multi-class
y_pred = [0, 2, 1, 3]
y_true = [0, 1, 2, 3]
acc = accuracy_score(y_true, y_pred)
print("Accuracy={}".format(acc))
acc = accuracy_score(y_true, y_pred, normalize=False)
print("Accuracy(normalize=False)={}".format(acc))
# Multi-label
y_pred = np.array([[0, 1, 1], [1, 1, 0]])
y_true = np.array([[1, 1, 0], [1, 1, 0]])
acc = accuracy_score(y_true, y_pred)
print("Accuracy={}".format(acc))
Accuracy=0.5
Accuracy(normalize=False)=2
Accuracy=0.5
Balanced accuracy score#
Balanced accuracy score 는 imblanced dataset 에서 성능을 측정할때 사용합니다. 이는 accuracy를 클래스의 비율에 따라 조절한 accuracy를 뜻하며 classification_report 결과에서 recall score 의 macro avg 값과 같습니다.
from sklearn.metrics import balanced_accuracy_score, classification_report
y_true = [0, 1, 0, 0, 1, 0]
y_pred = [0, 1, 0, 0, 0, 1]
acc = accuracy_score(y_true, y_pred)
print("Accuracy={}".format(acc))
bacc = balanced_accuracy_score(y_true, y_pred)
print("Balanced Accuracy={}".format(bacc))
report = classification_report(y_true, y_pred)
print("\nClassification Report\n{}".format(report))
Accuracy=0.6666666666666666
Balanced Accuracy=0.625
Classification Report
precision recall f1-score support
0 0.75 0.75 0.75 4
1 0.50 0.50 0.50 2
accuracy 0.67 6
macro avg 0.62 0.62 0.62 6
weighted avg 0.67 0.67 0.67 6
Confusion matrix#
Confusion matrix는 Predicted Class/Label 대비 True Class/Label 의 Count를 matrix 형태로 나타낸 것입니다.
Multi-class 의 경우 Column Index 가 Predicted Class이고, Row Index 가 True Class를 나타내고 Correct Count 값이 채워집니다.
Multi-label 의 경우는 Row 기준으로 각 Label 의 tp, tn, fp, fn 값이 채워집니다.
Actual/Predicted |
Predicted Negative |
Predicted Positive |
|---|---|---|
Actual Negative |
tn (true negative) |
fp (false positive) |
Actual Positive |
fn (false negative) |
tp (true positive) |
이는 각 경우에 대해서 Evaluation 함수를 계산하기 위한 값이 됩니다.
import numpy as np
from sklearn.metrics import confusion_matrix
from sklearn.metrics import multilabel_confusion_matrix
# Multi-class
y_pred = [0, 2, 1, 3, 2, 3, 1]
y_true = [0, 1, 2, 3, 1, 3, 1]
cm = confusion_matrix(y_true, y_pred)
print("Multi-class Confusion Matrix\n{}".format(cm))
# Multi-label
y_pred = np.array([[0, 1, 1], [1, 1, 0], [1, 1, 0]])
y_true = np.array([[1, 1, 0], [1, 1, 0], [1, 1, 0]])
mcm = multilabel_confusion_matrix(y_true, y_pred)
print("Multi-label Confusion Matrix\n{}".format(mcm))
Multi-class Confusion Matrix
[[1 0 0 0]
[0 1 2 0]
[0 1 0 0]
[0 0 0 2]]
Multi-label Confusion Matrix
[[[0 0]
[1 2]]
[[0 0]
[0 3]]
[[2 1]
[0 0]]]
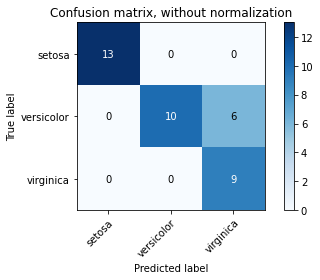
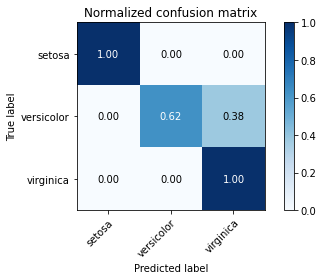
Multi-class 경우의 Confusion Matrix를 아래와 같이 시각화하여 나타내기도 합니다.
# 참조: https://scikit-learn.org/stable/auto_examples/model_selection/plot_confusion_matrix.html#sphx-glr-auto-examples-model-selection-plot-confusion-matrix-py
%matplotlib inline
import numpy as np
import matplotlib.pyplot as plt
from sklearn import svm, datasets
from sklearn.model_selection import train_test_split
from sklearn.metrics import confusion_matrix
from sklearn.utils.multiclass import unique_labels
# import some data to play with
iris = datasets.load_iris()
X = iris.data
y = iris.target
class_names = iris.target_names
# Split the data into a training set and a test set
X_train, X_test, y_train, y_test = train_test_split(X, y, random_state=0)
# Run classifier, using a model that is too regularized (C too low) to see
# the impact on the results
classifier = svm.SVC(kernel='linear', C=0.01)
y_pred = classifier.fit(X_train, y_train).predict(X_test)
def plot_confusion_matrix(y_true, y_pred, classes,
normalize=False,
title=None,
cmap=plt.cm.Blues):
"""
This function prints and plots the confusion matrix.
Normalization can be applied by setting `normalize=True`.
"""
if not title:
if normalize:
title = 'Normalized confusion matrix'
else:
title = 'Confusion matrix, without normalization'
# Compute confusion matrix
cm = confusion_matrix(y_true, y_pred)
# Only use the labels that appear in the data
classes = classes[unique_labels(y_true, y_pred)]
if normalize:
cm = cm.astype('float') / cm.sum(axis=1)[:, np.newaxis]
print("Normalized confusion matrix")
else:
print('Confusion matrix, without normalization')
print(cm)
fig, ax = plt.subplots()
im = ax.imshow(cm, interpolation='nearest', cmap=cmap)
ax.figure.colorbar(im, ax=ax)
# We want to show all ticks...
ax.set(xticks=np.arange(cm.shape[1]),
yticks=np.arange(cm.shape[0]),
# ... and label them with the respective list entries
xticklabels=classes, yticklabels=classes,
title=title,
ylabel='True label',
xlabel='Predicted label')
# Rotate the tick labels and set their alignment.
plt.setp(ax.get_xticklabels(), rotation=45, ha="right",
rotation_mode="anchor")
# Loop over data dimensions and create text annotations.
fmt = '.2f' if normalize else 'd'
thresh = cm.max() / 2.
for i in range(cm.shape[0]):
for j in range(cm.shape[1]):
ax.text(j, i, format(cm[i, j], fmt),
ha="center", va="center",
color="white" if cm[i, j] > thresh else "black")
fig.tight_layout()
return ax
np.set_printoptions(precision=2)
# Plot non-normalized confusion matrix
plot_confusion_matrix(y_test, y_pred, classes=class_names,
title='Confusion matrix, without normalization')
# Plot normalized confusion matrix
plot_confusion_matrix(y_test, y_pred, classes=class_names, normalize=True,
title='Normalized confusion matrix')
plt.show()
Confusion matrix, without normalization
[[13 0 0]
[ 0 10 6]
[ 0 0 9]]
Normalized confusion matrix
[[1. 0. 0. ]
[0. 0.62 0.38]
[0. 0. 1. ]]
Classification report#
Classification Report 에서는 Classification의 주요 성능 지표에 대한 결과를 테이블 형태로 출력해줍니다.
from sklearn.metrics import classification_report
y_true = [0, 1, 2, 2, 0, 1, 1]
y_pred = [0, 0, 2, 1, 0, 1, 1]
target_names = ['class 0', 'class 1', 'class 2']
print(classification_report(y_true, y_pred, target_names=target_names))
precision recall f1-score support
class 0 0.67 1.00 0.80 2
class 1 0.67 0.67 0.67 3
class 2 1.00 0.50 0.67 2
accuracy 0.71 7
macro avg 0.78 0.72 0.71 7
weighted avg 0.76 0.71 0.70 7
Precision, recall and F-measures#
Binary Classification 에서는 Precision, Recall, F-Measure 를 계산하기 위해 tp, fp, fn, tn 을 아래와 같이 정의하고 각 Metric을 계산합니다.
Predicted/Actual |
Actual Positive |
Actual Negative |
|---|---|---|
Predicted Positive |
tp (true positive) |
fp (false positive) |
Predicted Negative |
fn (false negative) |
tn (true negative) |
tp (true positive): Positive로 예측한 것이 True인 경우 (실제도 Positive 인 경우)
tn (true negative): Negative로 예측한 것이 True인 경우 (실제도 Negative 인 경우)
fp (false positive): Positive로 예측한 것이 False인 경우 (실제는 Negative 인 경우)
fn (false negative): Negative로 예측한 것이 False인 경우 (실제는 Positive 인 경우)
Precision
Positive로 예측한 것중에 실제 Positive 인 경우의 비율입니다.
\( \text{precision} = \frac{tp}{tp + fp} \)
Recall
실제 Positive 인 경우 중 Positive 로 예측한 것이 True 인 경우의 비율입니다.
\( \text{recall} = \frac{tp}{tp + fn} \)
F-measures
Precision과 Recall의 가중 조화 평균으로 모델의 성능을 평가합니다. 데이터에 따라 모델의 성능이 Precision이 더욱 중요한 케이스도 있고, Recall이 더욱 중요한 케이스도 있습니다. F-measure 에서는 F-beta 함수의 beta 값을 이용하여 각 케이스에 따라 Precision 과 Recall 의 중요도에 가중치를 적용하여 전체적 성능을 평가할 수 있습니다.
\( F_\beta = (1 + \beta^2) \frac{\text{precision} \times \text{recall}}{\beta^2 \text{precision} + \text{recall}} \)
예)
beta = 1: Precision과 Recall 의 중요도가 같음
beta = 2: Recall 이 Precision 보다 중요
beta = 0.5: Precision 이 Recall 보다 중요
from sklearn import metrics
y_pred = [0, 1, 0, 0]
y_true = [0, 1, 0, 1]
precision = metrics.precision_score(y_true, y_pred)
print("precision={}".format(precision))
recall = metrics.recall_score(y_true, y_pred)
print("recall={}".format(recall))
f1_score = metrics.f1_score(y_true, y_pred)
print("f1_score={}".format(f1_score))
f05_score = metrics.fbeta_score(y_true, y_pred, beta=0.5)
print("f0.5_score={}".format(f05_score))
f1_score = metrics.fbeta_score(y_true, y_pred, beta=1)
print("f1_score={}".format(f1_score))
f2_score = metrics.fbeta_score(y_true, y_pred, beta=2)
print("f2_score={}".format(precision, recall, f1_score,))
precision=1.0
recall=0.5
f1_score=0.6666666666666666
f0.5_score=0.8333333333333334
f1_score=0.6666666666666666
f2_score=1.0
Multi-Class/Label Classification 에서는 One vs the Rest 방식으로 각 Class의 Precision, Recall, F-Measure 값을 계산합니다. 그리고 각 Class 에 대한 Metric 의 Average 값은 주어진 옵션에 따라 아래 공식과 같이 계산됩니다.
“micro”: 전제 metric 에 대해서 sample-class pair 가 동등한 기여를 기반으로 계산됩니다. (sample-weight 의 결과는 제외)
“samples”: multi-label 문제에서만 사용되며, 각 sample를 위한 true(actual)/predicted class의 metric을 대신 사용합니다.
“macro”: 단순히 각 class의 가중치를 동등하게 binary metric의 평균으로 계산합니다.
“weighted”: mtuli-label 또는 class imbalance를 위해 각 class가 true data sample 내에 나타나는 비율로 가중된 binary metric의 평균을 계산합니다.
Selecting average=None: 각 class를 위한 score 배열이 반환됩니다.
각 Class/Label 의 Measure를 계산할때는 Average를 계산하여 산출하며, Average 계산방식은 아래 방식중 지정하여 계산이 가능합니다.
\(y \text{: the set of predicted } (sample, label) \text{ pairs }\)
\(\hat{y} \text{: the set of true } (sample, label) \text{ pairs }\)
\(L \text{: the set of labels}\)
\(S \text{: the set of samples}\)
\(y_s \text{: the subset of} y \text{with sample } s, \text{ i.e. } y_s := \left\{(s', l) \in y | s' = s\right\}\)
\(y_l \text{:the subset of } y \text{with label } l\)
\(\text{similarly, } \hat{y}_s \text{and} \hat{y}_l \text{are subsets of } \hat{y}\)
\(P(A, B) := \frac{\left| A \cap B \right|}{\left|A\right|} \text{for some sets } A \text{ and } B\)
\(R(A, B) := \frac{\left| A \cap B \right|}{\left|B\right|} \text{ (Conventions vary on handling } B = \emptyset \text{; this implementation uses } R(A, B):=0 \text{, and similar for } P\)
\(F_\beta(A, B) := \left(1 + \beta^2\right) \frac{P(A, B) \times R(A, B)}{\beta^2 P(A, B) + R(A, B)}\)
average |
Precision |
Recall |
F_beta |
|---|---|---|---|
“micro” |
\(P(y, \hat{y})\) |
\(R(y, \hat{y})\) |
\(F_\beta(y, \hat{y})\) |
“samples” |
\(\frac{1}{\mid S \mid} \sum_{s \in S} P(y_s, \hat{y}_s)\) |
\(\frac{1}{\mid S \mid} \sum_{s \in S} R(y_s, \hat{y}_s)\) |
\(\frac{1}{\mid S \mid} \sum_{s \in S} F_\beta(y_s, \hat{y}_s)\) |
“macro” |
\(\frac{1}{\mid L \mid} \sum_{l \in L} P(y_l, \hat{y}_l)\) |
\(\frac{1}{\mid L \mid} \sum_{l \in L} R(y_l, \hat{y}_l)\) |
\(\frac{1}{\mid L \mid} \sum_{l \in L} F_\beta(y_l, \hat{y}_l)\) |
“weighted” |
\(\frac{1}{\sum_{l \in L} \mid \hat{y}_l \mid} \sum_{l \in L} \mid \hat{y}_l \mid P(y_l, \hat{y}_l)\) |
\(\frac{1}{\sum_{l \in L} \mid \hat{y}_l \mid} \sum_{l \in L} \mid \hat{y}_l \mid R(y_l, \hat{y}_l)\) |
\(\frac{1}{\sum_{l \in L} \mid \hat{y}_l \mid} \sum_{l \in L} \mid \hat{y}_l \mid F_\beta(y_l, \hat{y}_l)\) |
“None” |
\(\langle P(y_l, \hat{y}_l) \mid l \in L \rangle\) |
\(\langle R(y_l, \hat{y}_l) \mid l \in L \rangle\) |
\(\langle F_\beta(y_l, \hat{y}_l) \mid l \in L \rangle\) |
from sklearn import metrics
y_true = [0, 1, 2, 0, 1, 2]
y_pred = [0, 2, 1, 0, 0, 1]
macro_precision = metrics.precision_score(y_true, y_pred, average='macro')
print("macro_precision={}".format(macro_precision))
micro_precision = metrics.recall_score(y_true, y_pred, average='micro')
print("micro_precision={}".format(micro_precision))
weighted_f1 = metrics.f1_score(y_true, y_pred, average='weighted')
print("weighted_f1={}".format(weighted_f1))
macro_fbeta = metrics.fbeta_score(y_true, y_pred, average='macro', beta=0.5)
print("macro_fbeta={}".format(macro_fbeta))
none_fbeta = metrics.precision_recall_fscore_support(y_true, y_pred, beta=0.5, average=None)
print("none_fbeta={}".format(none_fbeta))
macro_precision=0.2222222222222222
micro_precision=0.3333333333333333
weighted_f1=0.26666666666666666
macro_fbeta=0.23809523809523805
none_fbeta=(array([0.67, 0. , 0. ]), array([1., 0., 0.]), array([0.71, 0. , 0. ]), array([2, 2, 2]))
Receiver operating characteristic (ROC)#
ROC 커브는 이진 분류 시스템의 성능을 Positive 와 Negative 를 판별하는 Threshold 값을 다양하게 변화 시키면서 TPR(True Positive Rate)대 FPR(False Positive Rate)의 비율의 시각화하여 보여줌으로써 성능 차이를 판단할 수 있게 하는 방법입니다.
예를 들어, 두 이진 분류 모델 A, B가 존재할 경우 A 모델이 모든 Threshold 에서 ROC 의 값이 B보다 크다면 A 모델은 B 모델보다 성능이 좋다고 판단할 수 있습니다.
아래는 Multi-Class 를 Over vs the Rest 방식으로 이진 분류 모델로 학습하고 각 Class 에 대해서 테스트 데이터에 대하여 decision_function() 과 roc_curve() 함수를 이용하여 TPR, FPR, Threshold 값을 구하고 ROC Curve를 시각화화는 예제입니다.
# 참조: https://scikit-learn.org/stable/auto_examples/model_selection/plot_roc.html
import numpy as np
import matplotlib.pyplot as plt
from itertools import cycle
from sklearn import svm, datasets
from sklearn.metrics import roc_curve, auc
from sklearn.model_selection import train_test_split
from sklearn.preprocessing import label_binarize
from sklearn.multiclass import OneVsRestClassifier
from scipy import interp
# Import some data to play with
iris = datasets.load_iris()
X = iris.data
y = iris.target
# Binarize the output
y = label_binarize(y, classes=[0, 1, 2])
n_classes = y.shape[1]
# Add noisy features to make the problem harder
random_state = np.random.RandomState(0)
n_samples, n_features = X.shape
X = np.c_[X, random_state.randn(n_samples, 200 * n_features)]
# shuffle and split training and test sets
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=.5,
random_state=0)
# Learn to predict each class against the other
classifier = OneVsRestClassifier(svm.SVC(kernel='linear', probability=True,
random_state=random_state))
y_score = classifier.fit(X_train, y_train).decision_function(X_test)
# Compute ROC curve and ROC area for each class
fpr = dict()
tpr = dict()
roc_auc = dict()
for i in range(n_classes):
fpr[i], tpr[i], _ = roc_curve(y_test[:, i], y_score[:, i])
roc_auc[i] = auc(fpr[i], tpr[i])
# Compute micro-average ROC curve and ROC area
fpr["micro"], tpr["micro"], _ = roc_curve(y_test.ravel(), y_score.ravel())
roc_auc["micro"] = auc(fpr["micro"], tpr["micro"])
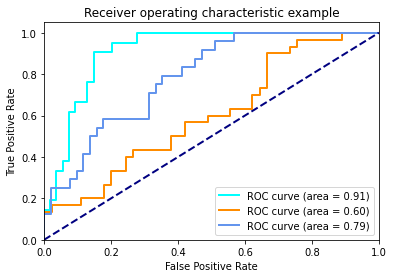
각 Class의 ROC Curve를 볼 수 있으며, Curve의 차이가 시각적으로 미비하다면, auc() 함수를 통해 구한 AUC (Area Under the Curve) 면적 값을 통해 하나의 값으로 비교 가능합니다.
plt.figure()
lw = 2
plt.plot(fpr[0], tpr[0], color='aqua',
lw=lw, label='ROC curve (area = %0.2f)' % roc_auc[0])
plt.plot(fpr[1], tpr[1], color='darkorange',
lw=lw, label='ROC curve (area = %0.2f)' % roc_auc[1])
plt.plot(fpr[2], tpr[2], color='cornflowerblue',
lw=lw, label='ROC curve (area = %0.2f)' % roc_auc[2])
plt.plot([0, 1], [0, 1], color='navy', lw=lw, linestyle='--')
plt.xlim([0.0, 1.0])
plt.ylim([0.0, 1.05])
plt.xlabel('False Positive Rate')
plt.ylabel('True Positive Rate')
plt.title('Receiver operating characteristic example')
plt.legend(loc="lower right")
plt.show()
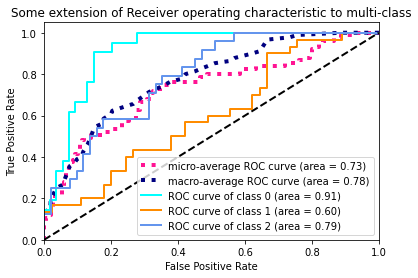
Multi-Class 분류 모델의 전체적인 성능을 비교하기 위해서는 각 Class 의 TPR과 FPR 값의 다양한 평균을 구하는 방식을 통해 하나의 값으로 비교가 가능하게 됩니다.
# Compute macro-average ROC curve and ROC area
# First aggregate all false positive rates
all_fpr = np.unique(np.concatenate([fpr[i] for i in range(n_classes)]))
# Then interpolate all ROC curves at this points
mean_tpr = np.zeros_like(all_fpr)
for i in range(n_classes):
mean_tpr += interp(all_fpr, fpr[i], tpr[i])
# Finally average it and compute AUC
mean_tpr /= n_classes
fpr["macro"] = all_fpr
tpr["macro"] = mean_tpr
roc_auc["macro"] = auc(fpr["macro"], tpr["macro"])
# Plot all ROC curves
plt.figure()
plt.plot(fpr["micro"], tpr["micro"],
label='micro-average ROC curve (area = {0:0.2f})'
''.format(roc_auc["micro"]),
color='deeppink', linestyle=':', linewidth=4)
plt.plot(fpr["macro"], tpr["macro"],
label='macro-average ROC curve (area = {0:0.2f})'
''.format(roc_auc["macro"]),
color='navy', linestyle=':', linewidth=4)
colors = cycle(['aqua', 'darkorange', 'cornflowerblue'])
for i, color in zip(range(n_classes), colors):
plt.plot(fpr[i], tpr[i], color=color, lw=lw,
label='ROC curve of class {0} (area = {1:0.2f})'
''.format(i, roc_auc[i]))
plt.plot([0, 1], [0, 1], 'k--', lw=lw)
plt.xlim([0.0, 1.0])
plt.ylim([0.0, 1.05])
plt.xlabel('False Positive Rate')
plt.ylabel('True Positive Rate')
plt.title('Some extension of Receiver operating characteristic to multi-class')
plt.legend(loc="lower right")
plt.show()
<ipython-input-10-5c21c2de8c65>:9: DeprecationWarning: scipy.interp is deprecated and will be removed in SciPy 2.0.0, use numpy.interp instead
mean_tpr += interp(all_fpr, fpr[i], tpr[i])